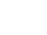Mejorando las habilidades en la lectura interpretada del antibiograma
2 CS de Marchena. Sevilla. (España).
EL ANTIBIOGRAMA: UTILIDAD EN LA PRÁCTICA CLÍNICA
El antibiograma es la prueba microbiológica que se realiza para determinar la sensibilidad antibiótica de una bacteria u hongo a uno o varios antimicrobianos. En el caso de la microbiología clínica, se trata de un germen previamente detectado en medios de cultivo y considerado agente causal de la patología que afecta al paciente.
Tras el diagnóstico clínico de una patología infecciosa hemos de instaurar una terapia empírica, basado en el conocimiento de los microorganismos causales y sus patrones de resistencia en el medio epidemiológico en el que tiene lugar. Este tratamiento empírico nos asegura una alta cobertura de los microorganismos más frecuentemente implicados o aquellos asociados a una mayor gravedad clínica.
En el caso de patologías en las cuales sea posible realizar una toma de muestra, siempre previa a la instauración del tratamiento antimicrobiano, contaremos con el apoyo diagnóstico que representan el cultivo y el antibiograma. La Tabla 1 enumera los métodos de determinación de resistencia para generar el antibiograma1-4.
Tabla 1. Métodos de determinación de resistencia. Mostrar/ocultar
El objetivo del antibiograma será facilitar la decisión terapéutica. Una vez conocido (con las técnicas clásicas unas 48h aproximadamente) nos permitirá seleccionar, entre los fármacos efectivos para el microorganismo, aquellos con un mejor perfil de efectos secundarios y un menor espectro antibiótico, instaurando así la terapia dirigida.
También nos va a servir de guía en las terapias secuenciales, aquellas en las cuales, siempre que se cumplan una serie de requisitos, podremos completar un tratamiento iniciado vía parenteral con otro administrado por vía oral.
También es importante recordar otros conceptos que pueden aparecer en el resultado del cultivo:
- Flora microbiana normal o habitual: se refiere al aislamiento de aquellos microorganismos colonizadores habituales de la zona corporal de la cual se ha tomado la muestra. Como ejemplo podríamos nombrar el aislamiento de Staphylococcus coagulasa negativos en una muestra cutánea.
- Flora contaminante: indica la presencia de microrganismos no procedentes de la zona corporal de la cual se ha tomado la muestra. Sería el caso del aislamiento de Propiobacterium acnes en una muestra de líquido cefalorraquídeo (LCR), procedente de la piel suprayacente, indicando una inadecuada desinfección cutánea.
PARÁMETROS FARMACOCINÉTICOS Y FARMACODINÁMICOS (PK/PD)
La interpretación del antibiograma se fundamenta en el análisis de los datos de sensibilidad, la detección de los mecanismos de resistencia, la farmacología del antimicrobiano y los resultados derivados de su utilización clínica5. La PK/PD varía dependiendo del régimen terapéutico, la formulación utilizada y la vía de administración. La respuesta del paciente tras la administración del antimicrobiano depende de muchas más variables que la acción frente a la bacteria, variables tanto farmacológicas como no farmacológicas6. El fármaco testado puede ser efectivo in vitro, pero nos debemos hacer varias preguntas como:
- ¿A qué dosis? ¿con qué intervalo?
- ¿Por cuál vía de administración?
- ¿Alcanzará las concentraciones adecuadas en el sitio concreto de infección?
- ¿Será potencialmente tóxico o será seguro para mi paciente?
- ¿Será potencialmente perjudicial para la ecología microbiana?
Dentro de los parámetros farmacológicos que afectan al tratamiento se encuentran la farmacocinética y la farmacodinamia como herramientas empleadas para optimizar la elección de las dosis de antimicrobianos:
- La farmacocinética (PK) describe las acciones del cuerpo sobre el fármaco administrado e incluye la absorción, distribución, metabolismo y excreción para definir la dosis sistémica.
- La farmacodinamia (PD) describe los efectos bioquímicos y fisiológicos del fármaco y su mecanismo de acción.
- Los análisis PK-PD se integran para definirla relación dosis-respuesta con el fin de identificar las pautas óptimas de dosificación.
La PK-PD antiinfecciosa es única en farmacología porque esta relación incluye los efectos del fármaco sobre el patógeno infeccioso y también sobre el huésped.
¿Qué es la concentración mínima inhibitoria (CMI)?
Es la mínima concentración de antibiótico que inhibe el crecimiento del MO (microorganismo) testado tras una incubación de 18-24 horas.
¿Cómo se mide la CMI? Los métodos más utilizados son los de dilución (Tabla 1), es decir, mediante la exposición del MO a distintas concentraciones creadas mediante diluciones seriadas de un antimicrobiano, determinando en cuál de ellas se deja de ver crecimiento (por turbidez). Es una medida in vitro cuyo valor no es absoluto (es el valor de una dilución) y depende de cada antibiótico y de cada microorganismo.
Clásicamente, un antibiótico se considera adecuado para tratar una infección causada por una bacteria cuando, administrado en dosis terapéuticas (es decir, no tóxicas), alcanza en el foco de infección concentraciones superiores a la CMI. En la práctica, una bacteria se considera sensible cuando las concentraciones plasmáticas (sangre) del antibiótico son cuatro veces superiores a la CMI, ya que las concentraciones plasmáticas se alcanzan en la mayoría de los tejidos, excepto algunos como el LCR, los huesos, la próstata o el globo ocular, en los que solo alcanzan concentraciones terapéuticas determinados antibióticos.
¿Qué son los puntos de corte clínicos? Representan los valores específicos de CMI según los cuales las bacterias se pueden asignar a las categorías clínicas (S-I-R) (Tabla 2)6. Recientemente, EUCAST ha redefinido la categoría Intermedia, de incertidumbre clínica, como Sensible Incrementando la exposición al antimicrobiano (tanto en dosis como en ajuste de intervalo) para determinados microorganismos y antibióticos7,8.
Tabla 2. Puntos de corte clínicos según CMI . Mostrar/ocultar
¿Cómo se determinan los puntos de corte? Mediante agencias oficiales con comités técnicos que van estandarizando y actualizando la información. En Europa se determinan mediante las normas de European Committee on Antimicrobial Susceptibility Testing (EUCAST), que determina los puntos de corte para dosis mínimas y máximas, así como los puntos de corte epidemiológicos (ECOFF).
¿Qué son los puntos de corte epidemiológicos (ECOFF)? Desde el punto de vista microbiológico, la distribución de los valores de concentración mínima inhibitoria (CMI) permite separar las poblaciones que no presentan/expresan mecanismos de resistencias (fenotipos salvajes) de aquellas que sí tienen dichos mecanismos (Figura 1). Estos puntos de corte pueden estimarse empleando procedimientos estadísticos, son conceptualmente invariables, no dependen de las dosis de antimicrobiano que puedan utilizarse y no necesariamente tienen que coincidir con los puntos de corte para definición de categorías clínicas. También nos sirven para identificar fenotipos de resistencia emergentes o bien raros o imposibles, por no haberse descrito (por ejemplo S. pyogenes resistente a penicilina). Resultan de utilidad para la lectura interpretada del antibiograma.
Figura 1. ECOFF y fenotipos de resistencia. Mostrar/ocultar
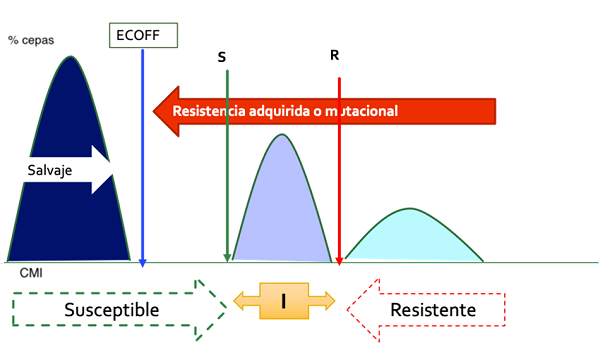
¿Para que sirven los parámetros PK/PD? Desde el punto de vista de la actividad PD, los antibióticos se clasifican en función del tipo de actividad antibacteriana y de la presencia de efecto post-antibiótico (EPA). Así, la actividad antibacteriana puede ser dependiente de la concentración si al aumentar la concentración del agente se produce una mayor eliminación del microorganismo, o dependiente del tiempo si la actividad antimicrobiana depende de la duración de la exposición del microorganismo al antibiótico. Con esto, los antibióticos se pueden clasificar en tres grandes grupos (Figura 2):
Figura 2. Clasificación farmacodinámica de antimicrobianos. Mostrar/ocultar
- Antibióticos con actividad concentración-dependiente (CD) y prolongado EPA. Para estos antibióticos, los parámetros relacionados con la eficacia son Cmax/CMI o el AUC24h/CMI. Estos antibióticos se utilizan a altas dosis, y el prolongado EPA permite utilizar intervalos de dosificación amplios (una dosis diaria). Ejemplo: utilización de aminoglucósidos en dosis única diaria (efecto pico terapéutico y utilización del EPA para evitar dosis valles tóxicas).
- Antibióticos con actividad tiempo-dependiente y efecto post-antibiótico mínimo o moderado. El objetivo de la terapia es conseguir una larga exposición al antibiótico. Para los antibióticos incluidos en este grupo, el tiempo durante el cual las concentraciones permanecen por encima de la CMI (T>CMI) es el parámetro relacionado con la erradicación bacteriana y la respuesta microbiológica. Este parámetro se denomina tiempo de eficacia. Cuanto menor es la semivida de eliminación, mayor es la frecuencia con la que hay que administrar estos antibióticos. Si la semivida es inferior a dos horas es difícil mantener un T>CMI por encima del 100% del intervalo de dosificación. En algunos casos, la perfusión continua es la forma más efectiva de administrar estos antibióticos, especialmente si se requiere un valor alto de T>CMI. Ejemplo: infusión extendida o continua de betalactámicos en infecciones graves o microrganismos sensibles cuando se incrementa la exposición.
- Antibióticos con actividad concentración- independiente y prolongado EPA. Al aumentar la concentración de estos antibióticos, la erradicación bacteriana aumenta solo ligeramente, pero se consigue una prolongada inhibición del crecimiento. El objetivo en estos casos es optimizar la dosis y el AUC24h/CMI es el parámetro relacionado con la eficacia (vancomicina, linezolid, tetraciclinas, clindamicina, azitromicina).
LECTURA INTERPRETADA DEL ANTIBIOGRAMA
La lectura interpretada consiste en un análisis fenotípico de los resultados de las pruebas de sensibilidad y se fundamenta en el conocimiento de los mecanismos de resistencia y su expresión fenotípica9,10. Su objetivo principal es evitar el posible fracaso terapéutico derivado del uso antimicrobiano cuando se expresan estos mecanismos de resistencia en la bacteria estudiada en el antibiograma. Esto tiene importantes implicaciones clínicas y epidemiológicas, ya que ayuda a los clínicos a poder indicar el tratamiento más adecuado a cada paciente a la vez que alerta, advierte y previene la aparición de resistencias antimicrobianas11.
En la lectura interpretada del antibiograma debe procesarse la información en función de los fenotipos obtenidos con el objetivo final de la detección del mecanismo de resistencia. El fenotipo de sensibilidad o de resistencia se define como el conjunto de datos obtenidos en el antibiograma para antibióticos de la misma familia o relacionados por mecanismos de actuación comunes o mecanismos de resistencia compartidos (Tabla 3). Para la detección de mecanismos de resistencia habituales se suelen utilizar drogas indicadoras que hacen extensiva su resistencia a otros miembros de esa familia de drogas. Se suele elegir para el cribado aquel ATB que presenta el mecanismo más obvio de resistencia.
Tabla 3. Resistencia antimicrobiana. Mostrar/ocultar
Fenotipos de resistencia desde el antibiograma (Tabla 4)
Tabla 4. Fenotipos de resistencia. Mostrar/ocultar
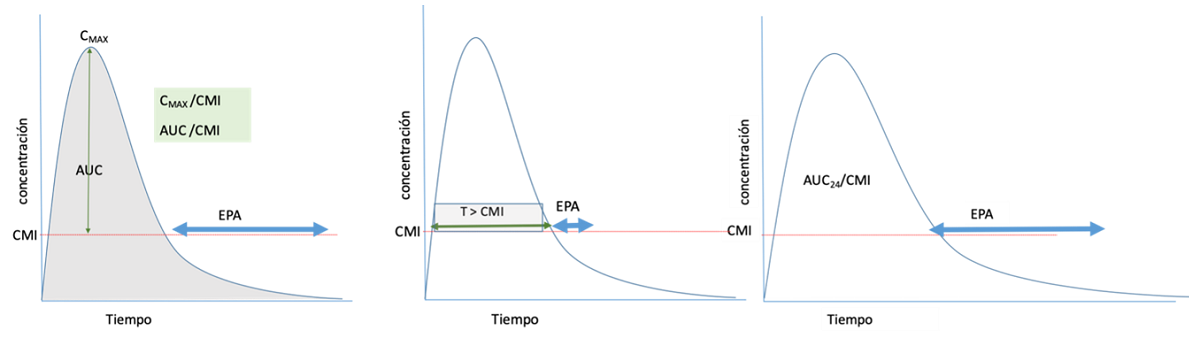
S. pneumoniae y resistencia a la penicilina12: disminución de la sensibilidad a amoxicilina debido a mutaciones en penicillin-binding proteins (PBP) que, dependiendo del tipo de mutación se expresará con mayor o menor nivel y, dependiendo del sitio de la infección, puede permitir utilizar penicilina a dosis elevadas con éxito terapéutico (Figura 3). La resistencia a amoxicilina no es dependiente de enzimas hidrolizantes (por tanto, No se beneficia de asociar inhibidores de beta-lactamasas como el ácido clavulánico).
Figura 3. CMI de S. pneumoniae a penicilina y puntos de corte. Mostrar/ocultar
S. pyogenes y resistencia a la penicilina: no hay descritas cepas con CMI >0,25 a penicilina (R), por lo que esto sería una definición de fenotipo imposible. S. pyogenes no es resistente a penicilina ni produce betalactamasas, (por tanto, no se beneficia de asociar inhibidores de beta-lactamasas como el ácido clavulánico).
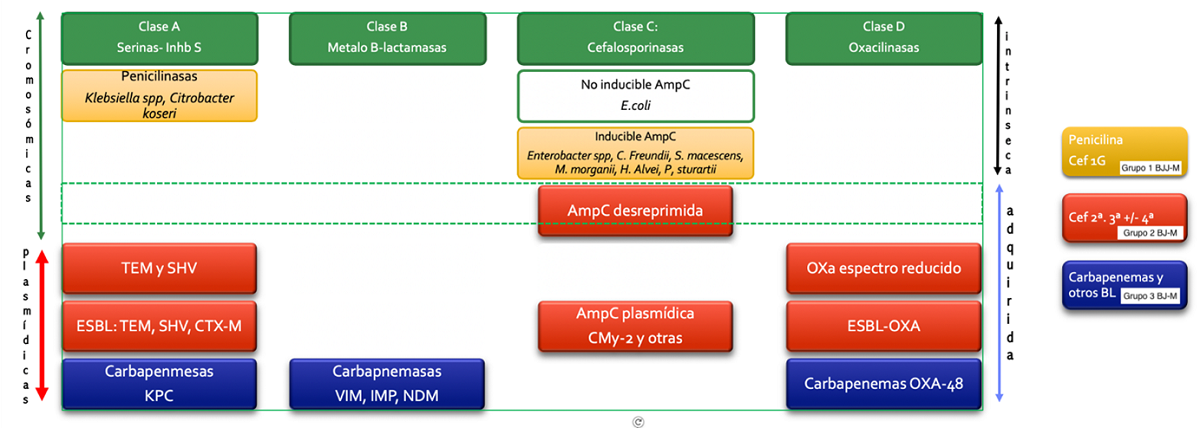
Resistencia a betalactámicos en enterobacterias13: el principal mecanismo de resistencia a betalactámicos en enterobacterias es el enzimático, por producción de betalactamasas. Las betalactamasas son enzimas que degradan el anillo betalactámico, actuando como mecanismo de resistencia natural en algunas bacterias. Las betalactamasas pueden clasificarse según su estructura de aminoácidos en cuatro clases moleculares, A-D, como sugirió por primera vez Ambler (Figura 4). Por otra parte, el sistema de clasificación de Bush-Jacoby-Medeiros divide las enzimas en varios tres grupos funcionales con subtipos de acuerdo con el perfil de su sustrato y sensibilidad a los inhibidores de la beta-lactamasa, como el ácido clavulánico. Según el grupo al que pertenezca la enterobacteria, puede presentar betalactamasas de forma natural (resistencia intrínseca), dato que es necesario conocer a la hora de interpretar el antibiograma. Fenotípicamente, las betalactamasas pueden ser de varios tipos (Tabla 5). La CMI a cefotaxima >1 mg/ml es un indicador de la posibilidad de producción de BLEE que el laboratorio debe confirmar. Las enterobacterias pueden asociar más de un tipo de mecanismo de resistencia adquirido, complicando la interpretación del antibiograma.
Figura 4. Betalactamasas clasificación de AMBLER y correspondencia con grupos de Bush, Jacoby-Medeiros. Mostrar/ocultar
Tabla 5. Betalactamasas. Mostrar/ocultar
CONCLUSIONES
La interpretación de un antibiograma es la categorización clínica del microorganismo en sensible, intermedio (sensible cuando se incrementa la exposición) o resistente. Esta nos va a informar de las posibilidades de éxito terapéutico con el antimicrobiano utilizado en su dosis habitual o incrementada.
Los valores numéricos de CMI no son comparables entre cada tipo de antimicrobiano, pues los puntos de corte que diferencian las categorías clínicas son distintos en cada uno de ellos.
Cuando realizamos una lectura interpretada del antibiograma estamos analizando el patrón de sensibilidades para así intentar predecir los mecanismos de resistencia que pudieran estar presentes. Conocer estos nos permitirá deducir la sensibilidad a algunos antibióticos no probados, así como en su caso la recategorización clínica de algunos otros.
BIBLIOGRAFÍA
- José A. García Rodríguez Rafael Cantón J. Elías García Sánchez Métodos Básicos de estudio de la sensibilidad a antimicrobianos. Recomendaciones de la SEIMC. En: Procedimientos en Microbiología Clínica [en línea] [consultado el 22/06/2021]. Disponible en: www.seimc.org/contenidos/documentoscientificos/procedimientosmicrobiologia/seimc-procedimientomicrobiologia11.pdf
- Doern GV. Antimicrobial Susceptibility Testing. J Clin Microbiol. 2011;49:S4-S4.
- March-Rosselló GA. Rapid methods for detection of bacterial resistance to antibiotics. Enferm Infecc Microbiol Clin. 2017;35:182-8.
- Charnot-Katsikas A, Tesic V, Love N, Hill B, Bethel C, Boonlayangoor S, et al. Use of the Accelerate Pheno System for Identification and Antimicrobial Susceptibility Testing of Pathogens in Positive Blood Cultures and Impact on Time to Results and Workflow. J Clin Microbiol. 2017;56:e01166-17.
- Breakpoint tables for interpretation of MICs and zone diameters. Version 11.0, 2021. En: The European Committee on Antimicrobial Susceptibility Testing [en línea] [consultado el 22/06/2021]. Disponible en: http://www.eucast.org
- Canut Blasco A, Aguilar Alfaro L, Cobo Reinoso J, Giménez Mestre MJ, Rodríguez-Gascón A. Análisis farmacocinético-farmacodinámico en microbiología: herramienta para evaluar el tratamiento antimicrobiano. Enferm Infecc Microbiol Clin. 2015;33:48-57.
- Kahlmeter G; the EUCAST Steering Committee. Redefining susceptibility testing categories S, I and R. En: The European Committee on Antimicrobial Susceptibility Testing [en línea] [consultado el 22/06/2021]. Disponible en: www.eucast.org/fileadmin/src/media/PDFs/EUCAST_files/EUCAST_Presentations/2018/EUCAST_-_Intermediate_category_-_information_for_all.pdf
- Aguilera-Alonso D, Martínez Campos L, Fernández Llamazares CM, Calvo C, Baquero-Artigao F; en representación del Grupo de Trabajo PROA-SEIP. Novedades en el antibiograma: “I” ya no significa sensibilidad intermedia. An Pediatr (Barc). 2021:S1695-4033(21)00182-X.
- Cantón R. Lectura interpretada del antibiograma: una necesidad clínica. Enferm Infecc Microbiol Clin. 2010;28:375-85.
- Gustinetti G, Cangemi G, Bandettini R, Castagnola E. Pharmacokinetic/pharmacodynamic parameters for treatment optimization of infection due to antibiotic resistant bacteria: a summary for practical purposes in children and adults. J Chemother. 2018;30:65-81.
- Soriano-García F. Aspectos farmacocinéticos y farmacodinámicos para la lectura interpretada del antibiograma. Enferm Infecc Microbiol Clin. 2010;28:461-6.
- Cillóniz C, García-Vidal C, Ceccato A, Torres A. Antimicrobial Resistance Among Streptococcus pneumoniae. En: Fong I., Shlaes D., Drlica K. (eds) Antimicrobial Resistance in the 21st Century. Emerging Infectious Diseases of the 21st Century. Springer, Cham; 2018.
- Navarro F, Miró E, Mirelis B. Lectura interpretada del antibiograma de enterobacterias. Enferm Infecc Microbiol Clin. 2010;28:638-45.